小笠原研TOP > カビ-T. reesei >基礎研究> 比較ゲノム解析
T. reeseiの比較ゲノム解析
ゲノム配列、遺伝子の発現量、表現型等を異なる菌株同士で比較することにより、「どのような変異が、セルラーゼ・ヘミセルラーゼの生産性を高めるという高機能化をもたらしたのか?」というバイオマス利用に向けた、根本的且つ重要な鍵となる新たな知見を得ることが可能になります。
近年、ゲノム配列を解読する装置(シークエンサー)の新世代タイプが身近なものとなりつつあります。それに伴って、さまざまな生物のゲノム配列が安価、且つ短時間で解読することが可能となり、ゲノム情報を元にした研究開発が活発化しています。
2005年に米エネルギー省(DOE)のJoint Genome Institute(JGI)によって、セルラーゼ高生産菌Trichoderma reesei の野生株(QM6a)の約34Mbpのゲノム配列が解読されました。T. reesei は、セルロース系バイオマス有効利用の観点から、数多くの高生産変異株が造成されています。しかし、それぞれの変異株について、変異点とセルラーゼ高生産性との関連は明らかになっていません。
当研究室では、T. reesei のセルラーゼ高生産変異株を数多く有しています。これらの変異株は、QM6a株の変異株であるQM9414を親株としてさまざまな変異が挿入されており、生育、セルラーゼ生産性等の表現型に大きな違いが見受けられます。
また、表現型の違いが、変異株の遺伝子配列の違い(多型性)に由来していると推測し、次世代シーケンサーSOLiDTM System(ABI社)によりいくつかの変異株のゲノムを解読し、配列を決定しています(データ未公開)。
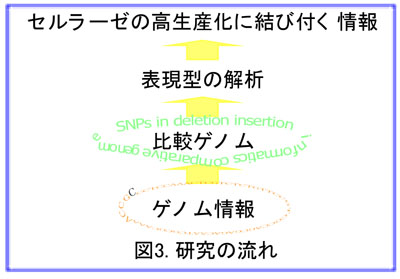
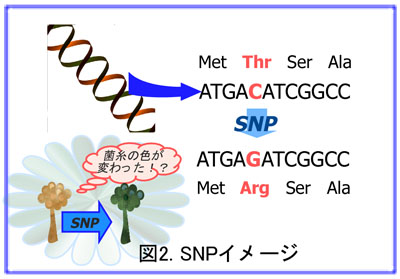
よって、当研究室では、野生株と変異株について、決定したゲノム配列情報を元に、一塩基置換(SNPs; 図2)*1、insertion(挿入)*2、in deletion(欠損)*3等の変異を対象とし、「セルラーゼ・ヘミセルラーゼの生産性向上」に資するであろう遺伝子をターゲットとし、図3比較ゲノム解析研究を進めています。
*1: 1塩基遺伝子多型性/Single nucleotide polymorphisms (SNPs)
もともとのゲノム配列から、アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の塩基の一つが別のものに置き換わっている変異。塩基が置換されることで、翻訳されるアミノ酸の種類が変わり、転写されるタンパク質等に変化がもたらされることがある。
*2: insertion(挿入)
もともとのゲノム配列に、新たに(複数の)塩基が挿入されている変異。
*3: in deletion(欠損)
もともとのゲノム配列から、(複数の)塩基が欠損している変異。
小笠原研TOP > カビ-T. reesei >基礎研究> 比較ゲノム解析
 研究紹介TOP
研究紹介TOP 基礎研究
基礎研究 比較ゲノム解析
比較ゲノム解析